protein-fa.htm
| Protein-Versuche: Protein-Fingerabdruck |
| Begriffe wie Genomanalysen oder "Genomics" haben mittlerweile Eingang in den normalen Wortschatz erhalten. Hierbei werden Chromosomen von verschiedener Organismen (u.a. Mensch) in ihrer Basenabfolge analysiert und katalogisiert. ( "HUGO"= humanes Genomprojekt). Diese Gensequenzen sind für die Wissenschaft jedoch so lange nutzlos, bis man ihre Genprodukte - die Proteine - kennt. Aber über das Zusammenspiel verschiedener Proteine in der Zelle, oder darüber, welche Proteine überhaupt exprimiert ("translatiert") werden, sagt "Genomics" nichts aus. Proteine sind die wichtigsten Bausteine des Lebens und übernehmen im Körper viele Funktionen: Enzyme, Membranproteine, Transportproteine, Hormone..). Die DNA hat hierbei lediglich die Aufgabe eines Informationsspeichers. Ein noch schwierigeres Vorhaben als die fast abgeschlossene Entschlüsselung des Genoms ist die Entschlüsselung des menschlichen Proteoms, der Gesamtheit aller menschlichen Proteine. Bisher sind jedoch nur ca. 5000 Proteine (von ca. 500.000) bekannt. Zu diesem Problem hat sich aktuell ein neuer Wissenschaftszweig, die "Proteomics" entwickelt. Hierbei wird die Gesamtheit der zu einem bestimmten Zeitpunkt (z.B. vor und nach einer Medikamenteneinnahme), unter ganz bestimmten Bedingungen (z.B. Stresseinwirkung) und an einem ganz bestimmten Ort (z.B. einer Tumorzelle) vorhandenen Proteine analysiert. Durch Vergleich des Protein-Profils einer kranken Zelle mit ihrer gesunden Nachbarzelle lassen sich oft erste Hinweise über Krankheitsursachen gewinnen. Die Entzifferung der Proteine verspricht also Heilungschancen für viele Krankheiten. In der Lebensmittelüberwachung ist die Bedeutung des Protein-Profils besonders gewachsen: Ist in Kalbsleberwurst auch drin, was auf dem Etikett steht? Nicht selten entdeckten Lebensmittelkontrolleure in Rindersalami nur Schweinefleisch oder Hühnerbrühe wurde mit billigerem Putenfleisch gestreckt. Angesichts der BSE-Gefahren wollen viele Verbraucher sicher sein, dass z.B. kein Rindfleisch in ihrer gekauften Wurstsorte enthalten ist. Auch der Nachweis gentechnisch veränderter Lebensmittel erhält in Zukunft große Bedeutung. Transgene Pflanzen mit artfremden Genen enthalten auch neue Proteine. Man wird in Zukunft z.B. gentechnisch veränderte Lebensmittel sehr einfach mit dieser Methode identifizieren können. Dazu wird das Vorhandensein eines Fremdproteins (z.B. eines Selektionsmarkerproteins) oder das Fehlen eines bestimmten endogenen Proteins untersucht. In der Evolutionsforschung spielt der Vergleich der Proteine ebenfalls eine große Rolle: Je ähnlicher die Proteinausstattung z.B. des Blutserums, desto ähnlicher muss auch die zugrundeliegende DNA sein, d.h. je geringer die genetische Distanz, desto enger die Verwandtschaft. |
| Die Erstellung eines Protein-Fingerabdrucks (z. B. Fleischsorten, Fruchtsäfte, Kartoffelsorten..) |
Da die Sequenz und Struktur eines jeden Proteins durch eine spezifische kodierende DNA-Sequenz festgelegt wird, können auch Protein-Fingerprints letzlich Aussagen über die genetische Ausstattung einer Art machen. Ähnlich wie beim DNA-Fingerprinting kann man durch Protein-Fingerprinting also evolutionäre Verwandtschaftsverhältnisse beschreiben. |
| Die SDS-Polyacrylamid-Gelektrophorese (SDS-PAGE) | |
Bei der SDS-Polyacrylamid-Gelelektrophorese werden Gemische verschiedener Proteine nach ihrem Molekulargewicht aufgetrennt. Die Proteinproben werden durch Zugabe des starken Detergens (=Seife) SDS (Natriumdodecylsulfat) und ß-Mercaptoethanol (=Reduktionsmittel) und anschließendes Erhitzen (95oC) denaturiert. Alle Proteine liegen im Anschluss an diese Denaturierung in einer ähnlichen, stäbchenartigen Form vor und sind durch das SDS stark negativ geladen. Das ß-Mercaptoethanol reduziert, d.h. spaltet eventuell vorhandene Disulfidbrücken innerhalb oder zwischen den Molekülen. | 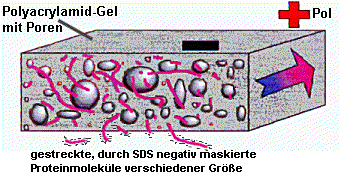 |
Legt man nach Auftrag der Proben auf das Gel ein elektrisches Feld an, wandern die Proteine zur Anode (+). Die Wanderungsgeschwindigkeit der Proteine hängt dabei fast nur von ihrem Molekulargewicht ab; ihre Eigenladung ist durch das SDS völlig überdeckt. Das SDS-PAGE kann man sich als eine Art Molekularsieb vorstellen, das aus langen Polymeren des Acrylamids besteht und durch Bisacrylamid quervernetzt wurde. Größere Proteine werden im Geflecht stärker zurückgehalten, wandern also langsamer. | |
| Material und Chemikalien: | |
| Thermoblock | Hitzedenaturierung | ||
| Stoppuhr | Zentrifuge, Elektrophorese | ||
| Elektrophoreseapparatur | Trennung der Proteine | ||
| Gel-Gießstand | Hilfe zum Gießen der Platten | ||
| Gilsonpipetten | |||
| Hamiltonpipette | Einfüllen der Proben in die Geltaschen | ||
| Schutzhandschuhe | |||
| Laemmli-Buffer + ßME | Lösungsmittel, Denaturierung der Proteine, Farbstoff, Fertiglösung Lagerung bei 4o C | SDS: H3C-(CH2)10-CH2-0S03- Na+ | |
| ßMercaptoethanol: HO-CH2-CH2-SH | |||
| Bromphenolblau | |||
| Laufpuffer: BioRad | Tris/Glycin/SDS-Fertiglösung: Leitfähigkeit | Lagerung bei Raumtemperatur | |
| Proteinmarker (fertig) | Zur Abschätzung der Molekülgrößen | Lagerung bei -20o C | |
| Trenngel-Lösung (fertig) | Rotiphorese Gel Fertig-Lösung (Acrylamid) | 8 mL | Lagerung bei 4o C ca. 1 Woche |
SDS-Lösung 10% (Sodiumdodecylsulfat) | 170 µL | ||
dest. Wasser | 5,3 mL | ||
| Tris 1,88 M, pH 8,8 (Puffer) | 3,5 mL | ||
| Starter | APS-Lösung 10% (Ammoniumperoxodisulfat): Starter | 100 µL | Zugabe bewirkt Polymerisation zu Polyacrylamid |
TEMED (Tetramethylethylendiamin) Radikalbildner | 10 µL | ||
| Sammelgel-Lösung (fertig) Im Bereich der Geltaschen zur besseren Auftrennung | Rotiphorese Gel 30 Lösung (Acrylamid) | 830µL | Lagerung ca. 1 Woche bei 4oC möglich |
| Tris 0,5 M, pH 6,8 (Puffer) | 500 µL | ||
| dest. Wasser | 3,6 mL | ||
| SDS-Lösung 10% (Sodiumdodecylsulfat) | 50 µL | ||
| Starter | APS-Lösung 10% (Ammoniumperoxodisulfat) : Starter | 40 µL | Zugabe bewirkt Polymerisation zu Polyacrylamid |
TEMED(Tetramethylethylendiamin) Radikalbildner | 5 µL | ||
| Isopropanol | Glätten des Gels | ||
| Coomassie- Färbelösung (fertig) | Ethanol 40% , Essigsäure7% , Coomassie Brilliant-Blue R250 0,4% dest. Wasser ad 100% | Raumtemperatur (Abzug) | |
| Coomassie- Entfärbelösung (fertig) | Ethanol 30% , Essigsäure10% dest. Wasser ad 100% | Raumtemperatur (Abzug) | |
| Durchführung: |
| A) Herstellung der verschiedenen Protein-Proben: Rindfleisch, Schweinefleisch, Putenfleisch und unbekannte Probe |
| 1) Stellen Sie die Proben der verschiedenen Fisch- bzw. Fleischarten bereit. Es genügt eine Menge, die einer Messerspitze entspricht (ca. 100 mg). Fettbestandteile sollten möglichst entfernt werden, da sie das Laufverhalten der Proteine in der Gelelektrophorese stark beeinträchtigen! Stellen Sie ebenfalls das Eppendorfgefäß mit dem Proteinmarker bereit. 2) Geben Sie zu jeder Fleischprobe 500 µL Laemmli Sample Buffer +ME und zum Proteinmarker 50 µL Laemmli Sample Buffer +ME und lassen Sie anschließend die Eppendorfgefäße 10 min bei 950C im Thermoblock inkubieren. Zur Vorbereitung der Proben sollten Einmalhandschuhe getragen werden, da die Eppendorf-Gefäße durch die Hitze aufspringen können! |
| 3) Alle 4 Proben werden 1min in der BioRad-Zentrifuge (bei 6000 rpm) zentrifugiert. Pipettieren Sie aus jedem Gefäß den Ãœberstand in neu beschriftete Eppendorfgefäße. 4) Dieser Ãœberstand mit dem denaturierten Proteingemisch wird weiterverwendet für die Gel- Elektrophorese. ( Bei Bedarf könnten die Proben jetzt zur Aufbewahrung eingefroren werden) |
| B) Gelelektrophorese | |||||
| 1) Herstellung des Trenngels: (ca. 17 mL, für 2 BioRad-Gele ausreichend) Es ist äußerst wichtig, beim folgenden Gießen der SDS-Gele Einmalhandschuhe zu tragen, da Acrylamid in seiner unpolymerisierten Form giftig ist! | |||||
5 mL der vorbereiteten Trenngellösung wird mit 30 µl der 10%igen APS-Lsung sowie 3 µl TEMED versetzt und zügig mit der 1000µl -Pipette in den Hohlraum zwischen die beiden Glasplatten pipettiert bis das FlÃüsigkeitsniveau bis zur Markierung angestiegen ist. Anschließend wird zu Glättung mit Isopropanol überschichtet (ca. 5 mm hoch). Nach ca. 10 bis 15 min ist das Trenngel polymerisiert. Das Isopropanol wird weggegossen, die Oberfläche vorsichtig mit dest. Wasser gespült und mit einem Filterpapier getrocknet. | |||||
| 2) Herstellung der Sammelgellösung (ca. 5 mL, für 2 BioRad-Gele ausreichend) Um das Sammelgel zu gießen, werden 4 mL der Sammelgellösung mit 30µL der 10%igen APS-Lösung sowie 4 µL TEMED versetzt und zügig mit der 1000µl -Pipette in den übriggebliebenen Hohlraum zwischen die beiden Glasplatten pipettiert. Anschließend wird sofort der Kamm eingesetzt (10 Zähne). Nach ca. 10 - 15 min ist auch das Sammel-Gel polymerisiert. Danach den Kamm vorsichtig entfernen! | |||||
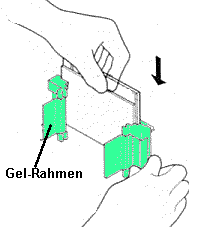
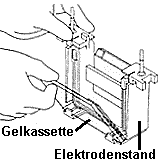
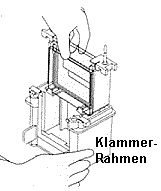
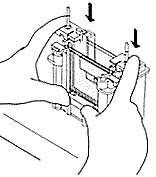
| 3) Zusammenbau der Gelapparatur und Elektrophorese: Der Gelrahmen wird wieder aus dem Gießstand entfernt und die beiden Glasplatten mit dem dazwischenliegenden Gel (=Gelkassette) ihrerseits aus dem Gelrahmen entnommen (optional wird an dieser Stelle mit einem Fertiggel begonnen). Die Gelkassette wird in den Elektrodenstand eingesetzt, so dass die kürzere Gelplatte nach innen (!) zeigt. Auf der gegenüberliegenden Seite wird optional eine weitere Gelkassette oder eine Blindplatte als Abdeckung eingesetzt (es können also 10 oder 20 Proben in einem Gellauf untersucht werden) Dieser Aufbau wird wie auf dem Bild beschrieben in den Klammerrahmen eingesetzt. Durch gleichzeitiges Herunterdrücken des Elektrodenstands und Umlegen der zwei Hebel nach innen (s. Abbildung) wird die innere Kammer zusammengesetzt. Die innere Kammer wird schließlich in den Pufferbehälter eingesetzt: Die innere Kammer und der Pufferbehälter werden mit Tris/Glycin/SDS-Puffer aufgefüllt, die äußere Kammer zwecks einfacherem Probenauftrag noch nicht ganz bis zum oberen Rand. Man benötigt insgesamt 1L Tris/Glycin/SDS-Puffer (10x Lösung von BioRad muss dazu 10fach verdünnt werden, d.h.100ml mit dest. Wasser auf 1l auffüllen). | |||||
|
|
|
| ||
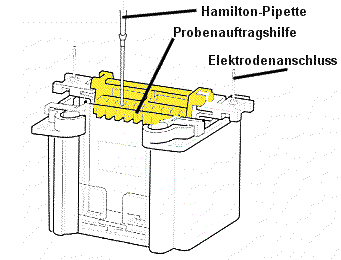
| 4) Probenauftrag: Die Probenauftragshilfe wird auf die Apparatur aufgesteckt. Mit einer Hamiltonspritze können jetzt die aufbereiteten Proben vorsichtig - d.h. ohne aus Versehen einen Teil der Flüssigkeit in Nachbar- Taschen zu bringen - in die Geltaschen aufgetragen werden. Es empfiehlt sich, auf den beiden Platten verschiedene Mengen zu versuchen: 5µL bzw. 10µL Reihenfolge protokollieren: Proteinmarker, Probe A bis X. Hamiltonpipette nach jeder Probe in der vorderen Pufferkammer spülen! Probenauftragshilfe entfernen! Der Deckel wird auf den Pufferbehälter aufgesetzt (Falschpolung ausgeschlossen) und an das Spannungsgerät angeschlossen. Die Elektrophorese wird 45 - 60 min bei 200 V durchgefhrt. Die Elektrophorese sollte abgeschaltet werden, wenn die blaue Bromphenolblau-Bande am unteren Gelrand angelangt ist. |
| ||||
| C) 1)Coomassie-Färbelösung: | |||||
Alle Bestandteile der Apparatur werden in umgekehrter Reihenfolge wieder auseinandergenommen, bis man die Gelkassette mit dem SDS-Gel vor sich liegen hat. Diese wird unter Zuhilfenahme eines Spatels oder grünen Keils von BioRad vorsichtig aufgehebelt und das Gel in die Coomassie-Färbelösung (ca.100 mL) überführt. Nachdem die Lösung in der Mikrowelle auf ca. 80oC gebracht wurde, wird für ca. 20 min unter ständigem Schwenken gefärbt. | |||||
2) Coomassie-Entfärbelösung: | |||||
| 3) Archivierung | |||||
| Zur Archivierung wird das Gel ca. 30 min in einer Ethanol/Glycerin-Lösung inkubiert, zwischen zwei in derselben Lösung (angefeuchtete Zellophan-Folien gelegt und in den Trockenrahmen (NOVEX) eingespannt. Das Gel ist nach ca. 12 h getrocknet und kann aus dem Rahmen entnommen werden. | |||||
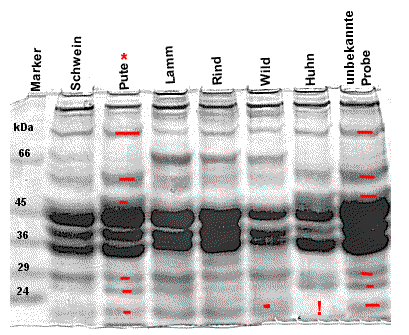
| D) Auswertung : "Erstellung eines Proteinfingerabdrucks (hier verschiedene Fleischproben)" | |||||
| Das Ergebnis des Versuchs "Erstellung eines Proteinfingerabdrucks (z.B. Fisch oder Fleisch)" sollte in etwa so aussehen wie in der Abbildung. Die zahlreichen Banden, die im SDS-PAGE sichtbar werden sollten, repräsentieren einen Teil der Proteine der untersuchten Fleischproben. Nicht alle Proteine können mit Coomassie angefärbt werden, da die Sensivität für gering exprimierte (=translatierte) Proteine beschränkt ist. Man erkennt, dass jede Fleischart (Pute, Rind, Schwein) ein ganz charakteristisches Muster an Proteinbanden zeigt. Bestimmte Banden kommen nur in einer der untersuchten Fleischsorten vor und können so auch zur Identifikation von Fleischsorten dienen (s. "unbekannte Probe). Auch in Fleischgemischen lässt sich so die Frage beantworten, ob z.B. Rindfleisch enthalten ist. (Vgl. auch Text in der Einleitung zu diesem Versuch) |
|
| Schülerzahl: 2 (max.4) Schülergruppen, da 2 Elektrophorese-Apparaturen mit max. 2 Gelplatten |
Lernvoraussetzungen: Sicherheit: |
| Zeitplanung: | Experiment: SDS-PAGE | ||||
Arbeitsschritt | |||||
Proteindenaturierung | A1) - A2) | Pipettieren, Inkubieren | 15' | ||
A3) - A4) | Zentrifugieren, Pipettieren | 5' | |||
Gelelektrophorese | B1) - B2) | Herstellung des Polyacrylamid-Gels | 45' | ||
B3) | Auftragen der Protein-Proben | 15' | |||
B4) | Elektrophorese | 60' | Aufräumen | ||
Färben/Entfärben |
| Anfärben der Protein-Banden | 20' | ||
Herauswaschen der Hintergrundfärbung | 30' | ||||
Archivierung | 20' | ||||
Auswertung | D) | 5' | |||
Gesamtdauer: | ca. 3,5h | ||||